15. 混合ガウスモデル(Gaussian Mixture Model, GMM)#
混合ガウスモデルは、以下の式で表される確率モデルで、多峰性のあるデータを表現するのに適している。
なんのことはない、複数のガウス分布の重ね合わせになっているだけだ。 ただし、ガウス分布の扱いの簡単さを残しつつ、より複雑な分布を表現できるようになる。
単一の正規分布との大きな違いは、その表現できる分布の形状にあるが、実用面における違いは、パラメータ推定にかかる難しさである。 確率分布の章で、単一の多次元正規分布の最尤推定の式を導出したが、混合ガウスモデルの場合は、最尤推定の式を解析的に解くことはできず、以降に述べるEMアルゴリズムを用いて、反復的にパラメータを推定する必要がある。
15.1. KLダイバージェンス#
EMアルゴリズムの説明をするために少し数学的な準備をしておく。
2つの確率分布 (P) と (Q) の間の差異を測る指標として、KLダイバージェンス(Kullback-Leibler divergence)がある。 これは、以下の式で定義される。
離散的な場合は、積分が和に置き換わる。 KLダイバージェンスは、非負であり、(P) と (Q) が同じ分布である場合にのみゼロになる。 また、非対称であり、一般に \(D_{KL}(P || Q) \neq D_{KL}(Q || P)\) である。つまり、KLダイバージェンスは距離の概念は満たさない。
離散的な場合を例に、シンプルな例を考えてみよう。
じゃんけんの手を出す確率がそれぞれ
Aさん: グー 0.5, チョキ 0.3, パー 0.2
Bさん: グー 0.2, チョキ 0.1, パー 0.7
Cさん: グー 0.4, チョキ 0.4, パー 0.2
であるとする。
このとき、ぱっと見で、AさんとCさんは似ているが、Bさんは違うな、と思う。 KLダイバージェンスを計算してみよう。 量を計算する場合は、特定の底(例えばネイピア数や2)を用いることに注意しよう。下では自然対数を用いている。
AさんとCさんのKLダイバージェンスが最も小さく、Bさんは他の2人とのKLダイバージェンスが大きいことがわかる。 今の場合は離散的だが、連続的な場合も、このKLダイバージェンスを用いて、2つの分布の差異を測ることができそうだ。
問題
以下の2つの1次元正規分布について、KLダイバージェンスを計算せよ。
15.2. EMアルゴリズム#
EMアルゴリズムは、EステップとMステップを交互に繰り返すことで、隠れ変数を含む確率モデルのパラメータを推定する手法である。 混合ガウスモデルだけでなく、潜在変数(latent variable) を含む様々な確率モデルに適用できる。
まずは一般論からスタートして、徐々に混合ガウスモデルに話を戻す流れで説明する。
15.2.1. 潜在変数#
潜在変数とは、観測されない変数のことで、データの生成過程に関与しているが、直接観測されない。 我々が観測できるのは、観測変数(observed variable)であり、背後には潜在変数が存在している、というのが基本的なアイデアである。
たとえば、学生の試験の点数(観測変数)があるとする。点数そのものは見えるが、その背後には「理解度」「集中力」「睡眠時間」 といった直接観測できない要因がある。これらが試験結果に影響しているが、教員が観測できるのは点数という観測データだけである。 この「理解度」や「集中力」のように、背後で観測データを生み出している隠れた要因が潜在変数である。
別の例として、たとえば100×100画素 RGB3色で表現されるたくさんの花の画像を考えてみよう。 これらの花の画像は30000次元の数値データ(ベクトル)として表現することができるわけだが、 花の画像は30,000次元の空間をランダムに埋め尽くしているわけではなく、同じ種類の花は「近い場所」に集まって分布しているはずである。花の種類によって分布の形も異なるだろう。 その背後には「花の種類」「花びらの形」「色の傾向」といった潜在変数が存在していると考えられる。
15.2.2. ELBO (Evidence Lower Bound; エビデンス下界)#
観測できる確率変数を \(x\)とし、潜在変数を \(z\) とする。 このとき、パラメータ\(\theta\)で表される確率モデルの対数尤度は、以下のように書ける。
\(z\)が離散的である場合を考えたが、連続的である場合は、和が積分に置き換わる:
また、上では簡単のため\(x,z\)が1つずつしかない場合を考えたが、実際には複数の観測データがある場合も同様である。
上の式の意味するところは、観測されたデータ\(x\)を説明する確率分布\(p_{\theta}\)を、パラメータ\(\theta\)を変化させながら、最も観測データをよく説明するように調整したい。 その際に、\(p_\theta(x)\)が、潜在変数\(z\)に関して周辺化(和または積分)されていることを意味する。
とくに、データセット \(\mathcal{D} = \{x^{(1)}, x^{(2)}, \ldots, x^{(N)}\}\) があった場合、対数尤度は以下のようになる。
この対数尤度を最大化したいわけだが、上の式はいわゆるlog-sumの形、対数の中に和が入っている形をしているため、直接的に最大化することが難しい。 これを解決するために、変分分布 (q(z)) を導入する。この変分分布は、潜在変数 (z) の近似的な分布を表すもので、任意に選ぶことができる(とはいえ、ガウス分布など簡単な分布を選ぶことが多い)。
この変分分布を用いて、対数尤度の下界を導出する。
最後の不等式は、Jensenの不等式を用いて導出される。この右辺がELBO (Evidence Lower Bound; エビデンス下界)である。
事後分布 \(p_{\theta}(z | x)\) と変分分布 \(q(z)\) のKLダイバージェンスを考えると、以下の関係が成り立つ。
ここから、
KLダイバージェンスは非負であるため、ELBOは対数尤度の下界であり、ELBOの最大化と事後分布と変分分布のKLダイバージェンスの最小化は同値であることがわかる。
15.3. EMアルゴリズムの概要#
上のELBO \(\mathcal{L}(q, \theta)\)は、変分分布\(q\)とパラメータ\(\theta\)の両方に依存している。 EMアルゴリズムは、以下の2つのステップを交互に繰り返すことで、ELBOを最大化する。
Eステップ: 現在のパラメータ\(\theta\)を用いて、潜在変数\(z\)の事後分布\(p_{\theta}(z | x)\)を計算する。
Mステップ: Eステップで得られた事後分布を用いて、ELBOを最大化するようにパラメータ\(\theta\)を更新する。
15.4. EMアルゴリズムを用いた混合ガウスモデルのパラメータ推定#
初めに、各ガウス分布のパラメータを \(\boldsymbol{\mu}_k\) (平均)、\(\boldsymbol{\Sigma}_k\) (共分散行列)、および混合係数 \(\pi_k\) (各ガウス分布の寄与度) とする。
Eステップ: 現在のパラメータを用いて、各データ点\(n\)がどのガウス分布\(k\)に属するかの確率を計算する。
\[ r_{nk} = \frac{\pi_k \mathcal{N}(\boldsymbol{x}_n | \boldsymbol{\mu}_k, \boldsymbol{\Sigma}_k)}{ \sum_j \pi_j \mathcal{N}(\boldsymbol{x}_n | \boldsymbol{\mu}_j, \boldsymbol{\Sigma}_j) } \]Mステップ: Eステップで得られた確率\(r\)を用いて、ガウス分布のパラメータ \(\boldsymbol{\mu}_k, \boldsymbol{\Sigma}_k, \pi_k\) を更新する。
\[ \boldsymbol{\mu}_k = \frac{1}{N_k} \sum_n r_{nk} \boldsymbol{x}_n, \quad \boldsymbol{\Sigma}_k = \frac{1}{N_k} \sum_n r_{nk} (\boldsymbol{x}_n - \boldsymbol{\mu}_k)(\boldsymbol{x}_n - \boldsymbol{\mu}_k)^\top, \quad \pi_k = \frac{N_k}{N} \]
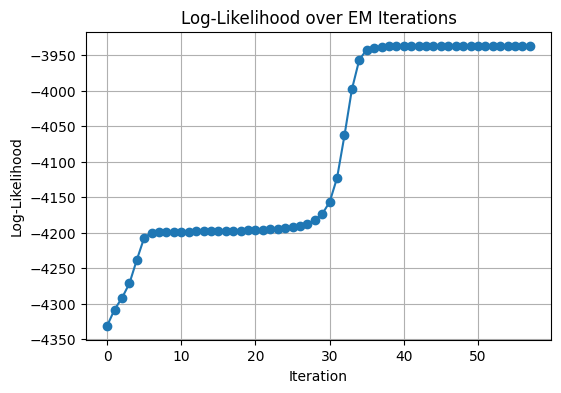
この逐次更新によって対数尤度の最大化を図り、収束するまでEステップとMステップを繰り返す。 対数尤度は以下のように表される。
Converged at iteration 57
Estimated mixing coefficients (phi): [0.3103679 0.30013538 0.38949671]
Estimated means (mu):
[[ 0.08871446 0.04564594]
[-3.08442868 3.07309071]
[ 3.05915799 3.15711097]]
Estimated covariances (sigma):
[[[ 1.01691342 0.38486941]
[ 0.38486941 0.98651172]]
[[ 1.44451192 0.28724678]
[ 0.28724678 0.91035083]]
[[ 1.09349783 -0.24274844]
[-0.24274844 1.27556857]]]
true_means = np.array([[0, 0], [3, 3], [-3, 3]])
true_covs = [
np.array([[1, 0.3], [0.3, 1]]),
np.array([[1, -0.2], [-0.2, 1.5]]),
np.array([[1.5, 0.4], [0.4, 1]])
]
true_weights = np.array([0.3, 0.4, 0.3])